Le syndrome respiratoire aigu sévère, également connu sous le nom de COVID-19 – dû au virus SARS-CoV-2 – se transmet par les gouttelettes respiratoires et par contact étroit.[1] La Lombardie et la plaine du Pô (Italie du Nord) ont été particulièrement touchées,[2] une région caractérisée par de fortes concentrations de particules fines, dont les effets néfastes sur la santé humaine sont déjà connus.[3] Les chiffres régionaux disponibles pour l'Italie au 12 avril montrent qu'environ 30 % des personnes actuellement positives vivent en Lombardie (environ 40 % si l'on considère l'ensemble des cas confirmés depuis le début de l'épidémie), suivie de l'Émilie-Romagne (13,5 %), du Piémont (10,5 %) et de la Vénétie (10 %).[2] Ces quatre régions de la plaine du Pô représentent 80 % du total des décès enregistrés en Italie et 65 % des admissions en soins intensifs.[2]
Une étude menée par l'École de santé publique de Harvard semble confirmer une association entre l'augmentation des concentrations de particules fines (PM) et les taux de mortalité liés à la COVID-19 aux États-Unis[4]. Dans des communications précédentes, nous avons émis l'hypothèse que le virus SARS-CoV-2 pourrait être présent sur les particules fines (PM) lors de la propagation de l'infection[5,6], ce qui concorde avec des données déjà disponibles.
disponible pour d'autres virus.[7-15] Cependant, la question du microbiome associé aux PM aéroportées, en particulier dans les environnements urbains, reste largement sous-étudiée,[16] et – à l'heure actuelle – personne n'a encore réalisé d'études expérimentales visant spécifiquement à confirmer ou à exclure la présence du SARS-CoV-2 sur les PM.
Nous présentons ici les premiers résultats des analyses que nous avons effectuées sur 34 échantillons de PM10 provenant d'un site industriel de la province de Bergame, collectés à l'aide de deux échantillonneurs d'air différents sur une période continue de 3 semaines, du 21 février au 13 mars.
Suivant la méthodologie décrite par Pan et al. en 2019 (pour la collecte, le dimensionnement des particules et la détection des virus aéroportés) [17], des échantillons de PM ont été prélevés sur des filtres en fibres de quartz à l'aide d'un échantillonneur d'air gravimétrique à faible volume (38,3 l/min pendant 23 h), conformément à la méthode de référence EN12341:2014 pour la surveillance des PM10. Les particules ont été retenues sur les filtres avec un taux de rétention typique de 99,9 %.Les aérosols retenus ont été correctement stockés et acheminés au laboratoire de génomique appliquée et comparative de l'université de Trieste. Compte tenu de la nature « environnementale » de l'échantillon, vraisemblablement riche en inhibiteurs d'ADN polymérases, nous avons procédé à l'extraction d'ARN à l'aide du kit Quick RNA pour micro-organismes fécaux du sol, adapté au type de filtres.[18] Un demi-filtre a été roulé, la face supérieure tournée vers l'intérieur.Dans un tube en polypropylène de 5 ml, avec les billes fournies dans le kit, nous avons introduit 1 ml de tampon de lyse initial. À partir de ce volume initial, nous avons obtenu environ 400 µl de solution, qui a ensuite été traitée selon les protocoles standard, donnant un éluat final de 15 µl. 5 µl de cet éluat ont ensuite été utilisés pour le test de dépistage du SARS-CoV-2. Compte tenu de l'origine particulière de l'échantillon, le kit qScript XLT 1-Step RT-qPCR ToughMix a été utilisé [19]. Les systèmes d'amplification étaient ceux du protocole développé par Corman et al., publié sur le site web de l'OMS [20].
Le test visait explicitement à confirmer ou à exclure la présence d'ARN du SARS-CoV-2 sur des particules. La première analyse, utilisant le gène E comme marqueur moléculaire, a donné un résultat positif impressionnant sur 15 filtres sur 16, même si, comme on pouvait s'y attendre, le Ct se situait entre 36 et 38 cycles.
Après cela, nous avons reproduit l'analyse sur 6 des filtres positifs (déjà positifs au « gène E ») en utilisant le « gène RtDR » comme marqueur moléculaire – qui est très spécifique du SARS-CoV-2 – atteignant 5 résultats significatifs de positivité ; des tests de contrôle pour exclure les faux positifs ont également été réalisés avec succès (Fig. 1).
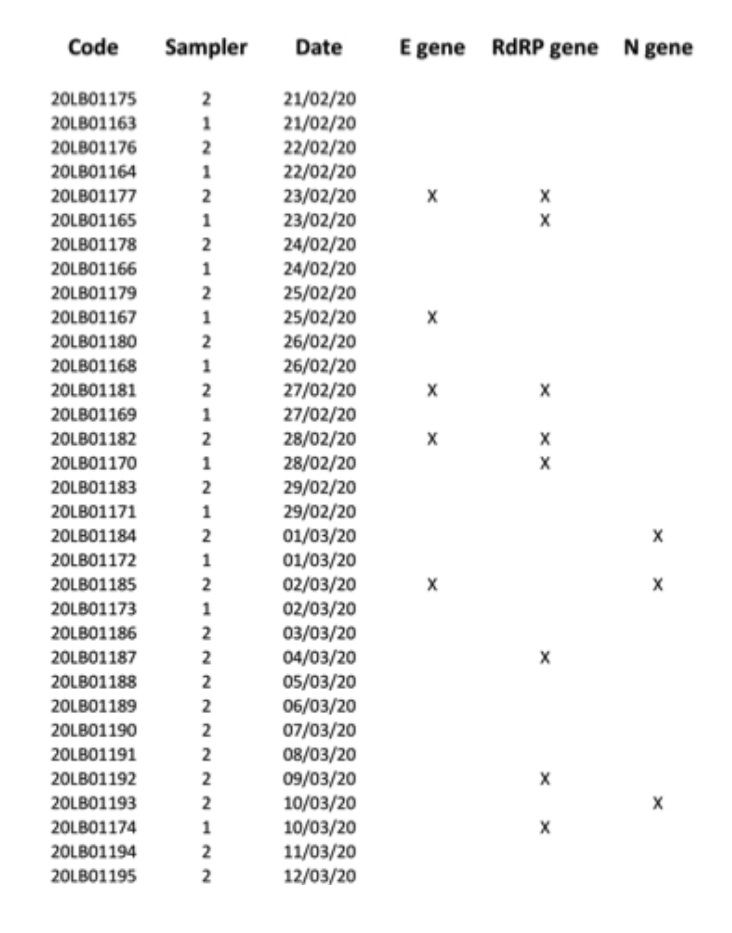
Afin d'éviter l'épuisement des stocks limités de matériel d'échantillonnage, les ARN extraits restants ont été acheminés vers le CHU local (l'un des centres cliniques agréés par le gouvernement italien pour les tests de diagnostic du SARS-CoV-2) pour la réalisation d'un second test en aveugle. Ce second laboratoire a analysé 34 extractions d'ARN pour les gènes E, N et RdRP, et a rapporté 7 résultats positifs pour au moins un des trois gènes marqueurs, la positivité étant confirmée séparément pour chacun des trois marqueurs (Fig. 2). Compte tenu de la nature de l'échantillon, et considérant que le prélèvement n'a pas été effectué à des fins de diagnostic clinique mais pour des analyses de pollution environnementale (et sachant également que les filtres ont été conservés pendant au moins quatre semaines avant les analyses de génétique moléculaire),Suite au confinement en Italie, nous avons pu démontrer de manière convaincante la présence d'ARN viral du SARS-CoV-2 en détectant le gène RtDR, hautement spécifique, sur 8 filtres. Cependant, faute de matériel supplémentaire provenant de ces filtres, nous n'avons pas pu répéter un nombre suffisant de tests pour obtenir simultanément la positivité des 3 marqueurs moléculaires.
Il s'agit des premières preuves préliminaires de la présence d'ARN du SARS-CoV-2 sur les particules en suspension dans l'air extérieur. Ceci suggère que, dans des conditions de stabilité atmosphérique et de fortes concentrations de particules, le SARS-CoV-2 pourrait former des agrégats avec ces particules et, en réduisant leur coefficient de diffusion, favoriser la persistance du virus dans l'atmosphère. D'autres confirmations de ces résultats préliminaires sont attendues.Les données sont en cours d'analyse et devraient inclure une évaluation en temps réel de la vitalité du SARS-CoV-2 ainsi que de sa virulence lorsqu'il est adsorbé sur des particules fines. À l'heure actuelle, aucune conclusion ne peut être tirée quant à la corrélation entre la présence du virus sur les particules fines et la progression de l'épidémie de COVID-19. Parmi les autres points à examiner figurent les concentrations moyennes de particules fines.nécessaire pour un éventuel « effet de rappel » de la contagion (au cas où il serait confirmé que les PM pourraient agir comme un « vecteur » pour les noyaux de gouttelettes virales), voire pour la possibilité théorique d’une immunisation consécutive à des expositions à des doses minimales à des seuils inférieurs de PM.
Fig. 1 Courbes d'amplification des gènes E (A) et RdRP (B) : les lignes vertes représentent les filtres testés ; les lignes croisées, les lignes droitesreprésente les extractions du filtre de référence ; les lignes rouges représentent l’amplification des échantillons positifs.
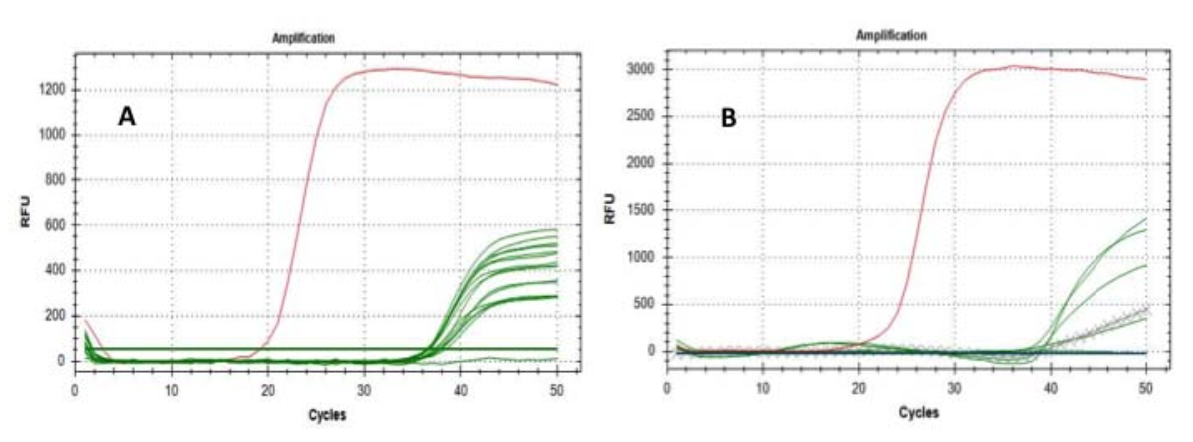
Figure 2. Résultats positifs (marqués d'une croix) pour les gènes E, N et RdRP obtenus pour les 34 échantillons PM10.filtres testés dans la seconde analyse parallèle.
 Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
1. Département de chimie industrielle, Université de Bologne, Viale del Risorgimento – 4, I-40136, Bologne, Italie
e-mail: leonardo.setti@unibo.it
2. Centre interdépartemental de recherche industrielle « Sources renouvelables, environnement, croissance bleue, énergie »,
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Département de biologie, Université « Aldo Moro » de Bari, Bari, Italie
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Département des sciences chimiques et pharmaceutiques, Université de Trieste, Trieste, Italie
e-mail: barbierp@units.it
5. Division de recherche environnementale, TCR TECORA, Milan, Italie
e-mail: mariagrazia.perrone@tcrtecora.com
6. Département des sciences de la vie – Université de Trieste, Trieste, Italie
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. Division de médecine de laboratoire, hôpital universitaire Giuliano Isontina (ASU GI), Trieste, Italie
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Société italienne de médecine environnementale (SIMA), Milan, Italie
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Département des sciences et politiques environnementales, Université de Milan, Milan, Italie
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Auteur correspondant :
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Références
1. Organisation mondiale de la santé, Modes de transmission du virus responsable de la COVID-19 : implications pour les recommandations en matière de précautions de prévention et de contrôle des infections, Note scientifique ; disponible sur : https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (29 mars 2020)
2. Ministère italien de la Santé, bulletin quotidien sur l'épidémie de Covid-19 en Italie, disponible à l'adresse http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. AEE, Agence européenne pour l'environnement, Rapport sur la qualité de l'air en Europe 2019 ; n° 10/2019 ; Agence européenne pour l'environnement : Copenhague, Danemark, disponible à l'adresse : https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, « Exposition à la pollution atmosphérique et mortalité liée à la COVID-19 aux États-Unis », disponible à l’adresse : https://projects.iq.harvard.edu/files/covid-pm/files/pm_and_covid_mortality.pdf
5. Société italienne de médecine environnementale (SIMA), Document de position sur les particules fines et la COVID-19,
Disponible à l'adresse suivante : http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ENG.pdf
6. Setti L., Passarini F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Is There a Plausible Role for Particulate Matter in the spread of COVID-19 in Northern Italy ?, BMJ Rapid Responses, 8 avril 2020, disponible sur : https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Génération de particules fines fécales contaminées par le virus de la grippe aviaire (VGA) : détection du génome et de l’infectiosité et calcul de l’immission. Veterinary Microbiology. 139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. La transmission par voie aérienne pourrait avoir joué un rôle dans la propagation des épidémies de grippe aviaire hautement pathogène de 2015 aux États-Unis. Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Évaluation de l'impact des épisodes de poussière sur l'incidence de la rougeole dans l'ouest de la Chine. Atmospheric Environment. 157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI. Un modèle intégré pour prédire la propagation atmosphérique du virus de la fièvre aphteuse. Epidemiol. Infect., 124, 577–590 (2000)
11. Glostera, J., Alexandersen, S. Nouvelles orientations : Transmission aéroportée du virus de la fièvre aphteuse Environnement atmosphérique, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA. Taux de dépôt des virus et des bactéries au-dessus de la couche limite atmosphérique. The ISME Journal. 12, 1154-1162 (2018)
13. Qin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H., Knight, R., Gilbert, JA, Zhu, TF. Étude longitudinale du microbiome associé aux particules fines dans une mégapole. Genome Biology. 21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. La transmission par voie aérienne pourrait avoir
a joué un rôle dans la propagation des épidémies de grippe aviaire hautement pathogène de 2015 aux États-Unis. Sci
Réf. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Évaluation de l'impact des épisodes de poussière sur l'incidence de la rougeole dans l'ouest de la Chine. Atmospheric Environment. 157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J., Lang, J., Tian, G., Jiang, J., Zhu, TF. Extraction d'ADN optimisée et séquençage métagénomique des communautés microbiennes aéroportées. Nat. Protoc. 10, 768-779 (2015)
17. Pan, M., Lednicky, JA, Wu, C.-Y., Collecte, dimensionnement des particules et détection des virus aéroportés. Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ltd, description du produit, disponible sur : https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, description du produit, disponible sur : https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK et Mulders, DG (2020).
Détection du nouveau coronavirus 2019 (2019-nCoV) par RT-PCR en temps réel. Eurosurveillance, 25(3), disponible à l'adresse : https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Article original : https://doi.org/10.1101/2020.04.15.20065995
Date de publication : 18 avril 2020








